Há uma tentativa natural de alguns colegas brasileiros de mudar o nome para algo mais nacional. Os candidatos à tradução são "editoração" e "processamento". No entanto, "splicing" tem revivido a história de alguns outros termos como "feedback" e "crossing-over", por exemplo.
O processo foi anunciado cientificamente em 1977. Tanto tempo sem conhecê-lo deve-se ao excesso de trabalhos, no início da biologia molecular, com os procariotos. Pouca coisa era investigada em eucariotos. Acontece, que o "splicing" só ocorre em células eucarióticas.
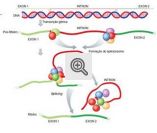
O "splicing" consiste na remoção de fragmentos de um RNA recém-sintetizado (chamado pré-RNA). Isso ainda se passa no núcleo (embora já haja indícios que possa se dar no citoplasma de neurônios). Esses pedaços removidos são chamados de íntrons. Os pedaços que persistem no RNA maduro são chamados de éxons. A remoção exige um complexo formado por 5 espécies de RNA e mais 50 proteínas. É chamado de spliceossomo, a máquina de excisão. Essa monstruosa parafernália reconhece início e fim dos íntrons, depois os unem, formando uma alça que então é cortada. Um íntron pode ter de 10 a até 100 mil nucleotídeos! O início e o fim do íntron ainda é um mistério. Mesmo com auxílio de computadores, as sequências de reconhecimento pelo complexo ribonucleoprotéico (o spliceossomo) ainda é mal compreendido.
Cerca de 60% dos genes eucarióticos parecem sofrer "splicing". Não é uma unanimidade, como se vê. No início, o mecanismo foi visto como algo desnecessário. Hoje, olhamos para ele como uma grande justificativa para o alto potencial que os genomas eucarióticos apresentam. Como o nosso, por exemplo. Mas qual é essa vantagem?
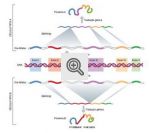
O Projeto Genoma Humano identificou aproximadamente 24 mil genes. Cada um deles tem potencial para a produção de um RNA, seja mensageiro (a imensa maioria), transportador ou ribossômico. Acontece o nosso proteonoma (o acervo protéico) apresenta mais de 100 mil proteínas! Como se o número de informações (os genes) é bem menor? A resposta parece estar no "splicing". Observe o esquema abaixo.
Percebeu que o mesmo gene foi transcrito da mesma forma mas resultou em duas moléculas de RNA maduros diferentes? Cada uma delas resultará em dois polipeptídios distintos que poderão ter funções distintas em momentos diferentes, ou em células diferentes. Chama-se de "splicing" alternativo e parece ser o segredo do sucesso evolutivo do mecanismo. Senão seria um desperdício metabólico inexplicável. Bom, mas poderia ser essa a explicação. Às vezes ciência é um pouco confusa mesmo...